ac4C RNA乙酰化是在RNA ac4C修饰酶的作用下,使N4位乙酰胞嘧啶发生乙酰化的一种保守的化学修饰(N4-acetylcytidine)。早期研究发现该修饰存在于真核生物中丝氨酸及亮氨酸tRNA和18S rRNA上,导致Watson-Crick碱基热稳定性增加,调控蛋白合成中的编码准确性;近期研究发现ac4C分布在人类转录组中,大多数位点出现在编码序列(CDS)内,并且通过改善mRNA的稳定性和翻译促进靶基因表达。NAT10催化的ac4C通过促进mRNA的稳定性,成为编码转录组中一个重要的转录后调节因子。然而,它在哺乳动物发育中的作用尚不清楚。
云序客户北京大学基础医学科学院李扬科研团队通过云序生物提供的acRIP-seq、LC-MS/MS等技术,发现核心多能性调节因子OCT4是人胚胎干细胞(hESC)自我更新维持过程中独特且显著的NAT10应答的ac4C修饰靶基因,揭示了NAT10在维持hESC自我更新中的作用。该研究成果以“NAT10-mediated N4-acetylcytidine mRNA modification regulates self-renewal in human embryonic stem cells”为题发表在《Nucleic Acids Research》杂志上。

发表期刊:Nucleic Acids Research
影响因子:14.9/Q1
发表时间:2023年7月27日
期刊ISSN:0305-1048
研究方法:acRIP-seq、acRIP-qPCR、LC-MS/MS等
研究思路
研究内容
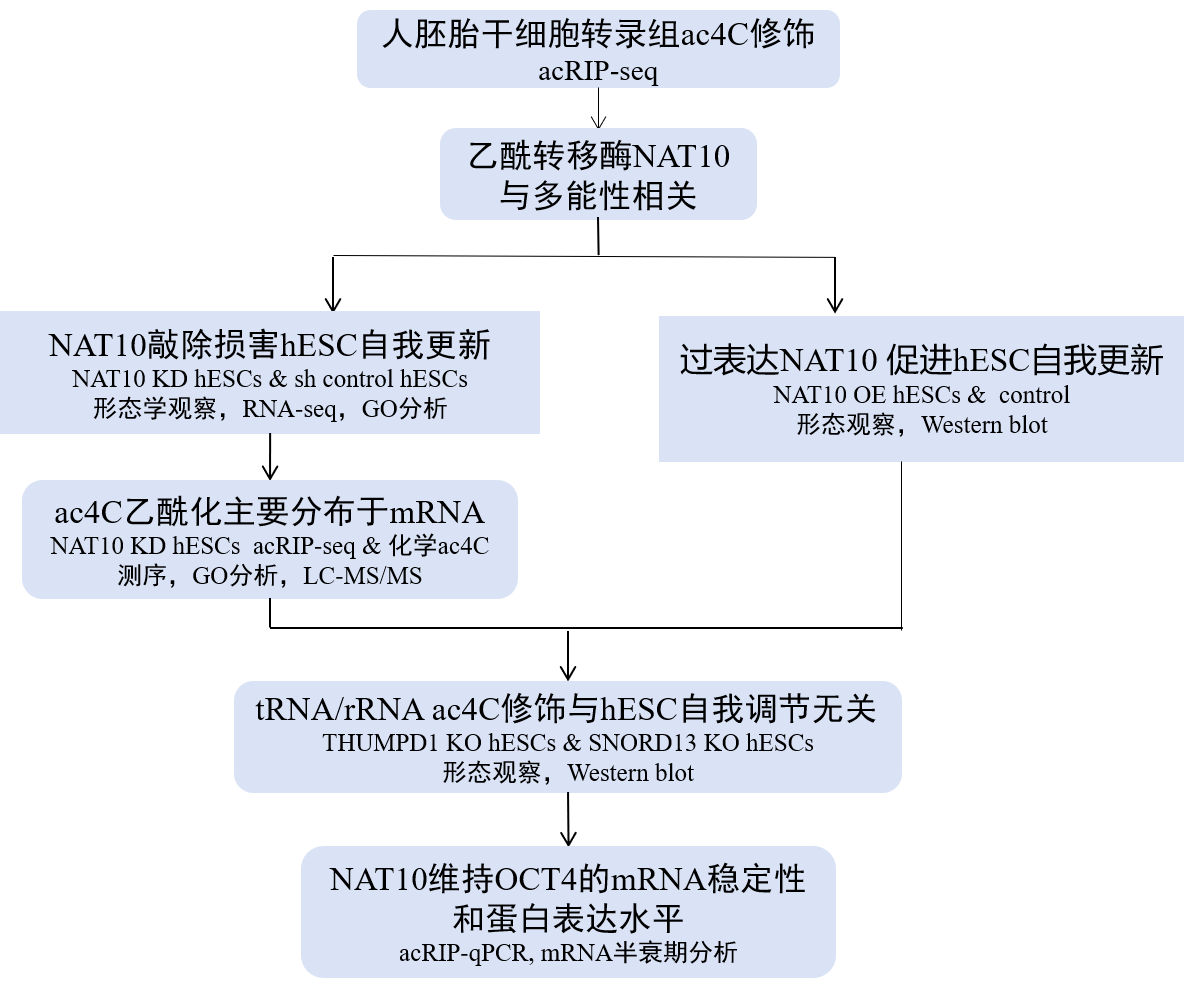
1、高通量测序显示ac4C修饰在hESC转录组中普遍存在
作者通过acRIP-seq定位了ac4C修饰在野生型(WT)hESC上的位置,观察到ac4C峰在编码序列(CDS)中富集,也有相当一部分会在3’-UTRs中富集。在具有ac4C修饰的mRNA中,我们观察到编码核心多能性调节因子OCT4(POU5F1)、SOX2和NANOG的mRNA始终被ac4C修饰,此外,还检测到其他与多能性相关的基因转录本具有ac4C修饰。
2、乙酰转移酶NAT10的表达与多能性呈正相关
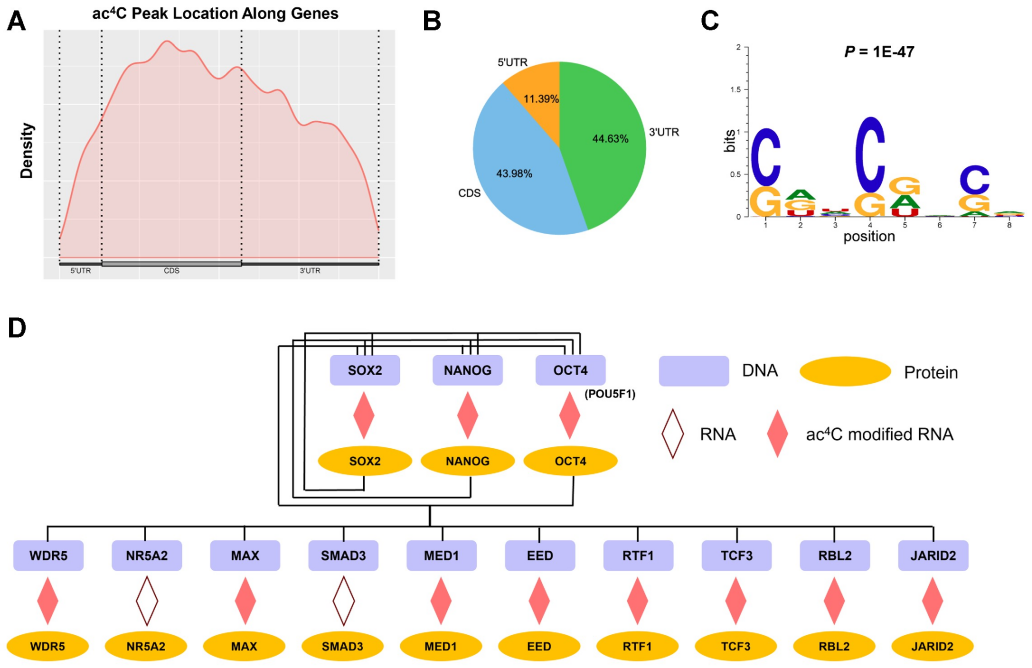
为了了解ac4C修饰在多能性维持中的作用,作者重点研究了NAT10,这是人类中唯一已知的ac4C乙酰转移酶,其表达水平直接影响ac4C在RNA上的形成。作者首先研究了NAT10在人类早期胚胎发育过程中的表达模式,发现NAT10在外胚层中高表达,但在三胚层发育过程中表达显著下调。随后通过EB(胚状体)形成进行自发分化实验,发现在自发分化过程中,mRNA和蛋白水平上NAT10的表达显著下降,与OCT4的表达趋势相似。定向分化检测显示在早期分化过程中NAT10和OCT4下调,分化标志物SOX1和SOX17在mRNA和蛋白水平上均上调。

3、NAT10的扰动会损害hESC的自我更新,并促进hESC的早期分化
作者构建了NAT10 KD hESCs(RNAi敲低NAT10)和sh control hESCs(shRNA 慢病毒对照)两种类型细胞,在相同条件下培养后,KD细胞比sh对照细胞表现出更少、更小的菌落,增值效率也显著降低。用Western blot和RT-qPCR方法检测多能性标记物和发育调控因子的基因表达,相比于sh control hESCs,NAT10 KD hESCs显示核心多能性调节因子OCT4、NANOG、SOX2的表达减少,而三胚层的发育调节因子表达增加。为了更全面地分析NAT10敲低导致的hESCs基因表达的改变,作者对NAT10 KD和sh control的hESCs进行了高通量RNA测序,结果显示,NAT10基因的敲除导致2455个基因表达下调,1548个基因表达上调。对NAT10 KD hESCs中下调的差异表达基因(DEGs)的GO-BP功能富集分析显示,部分基因在多细胞生物信号通路、第二信号通路介导的信号通路和多种信号通路中富集。对NAT10 KD hESCs中上调的DEGs进行分析,显示出与形态发生、发育和分化相关的显著功能,如肾脏形态发生、内胚层发育和内胚层细胞分化。综上所述,结果表明NAT10敲除的干扰损害了hESCs的自我更新,并促进了早期分化。
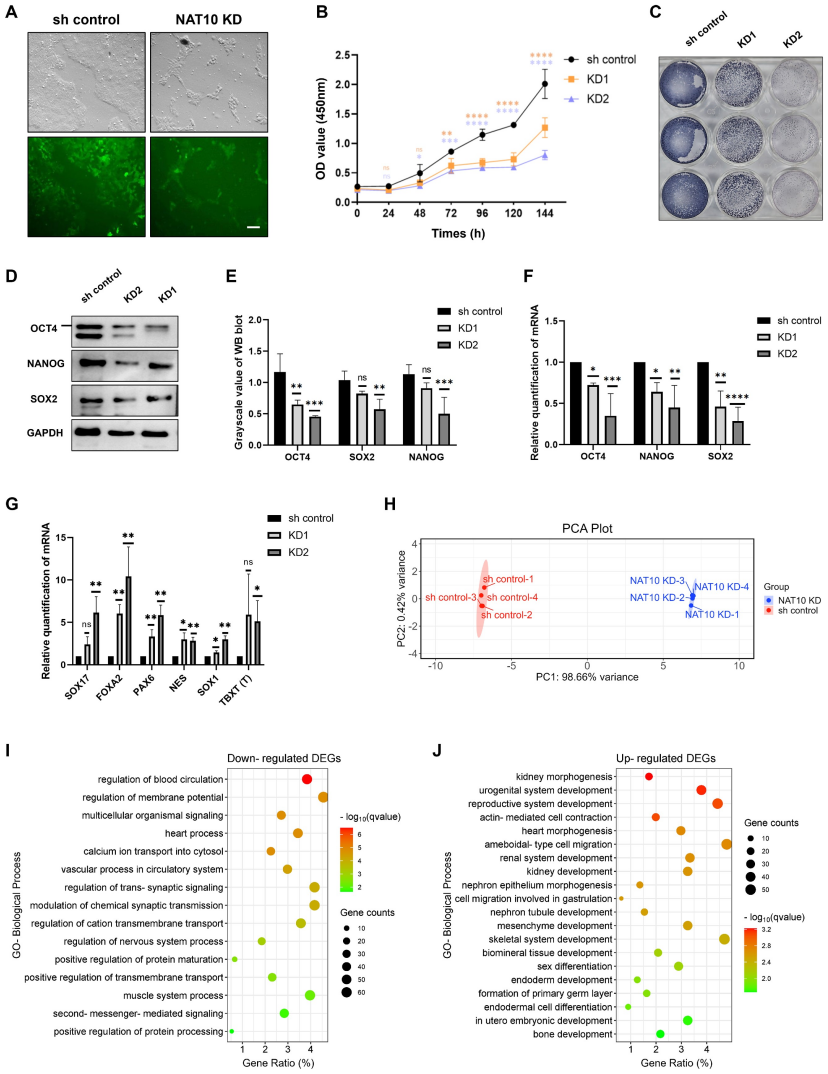
4、过表达NAT10可促进hESC的自我更新
作者生成过表达细胞系(NAT10 OE hESCs),以研究NAT10在hESCs中是否为自我更新的调节因子。实验结果显示,NAT10 OE hESCs与对照组保持相同未分化形态,NAT10 OE hESCs的增殖率明显高于对照hESCs。对NAT10过表达组和对照组中核心多能标记物的Western blot检测结果发现,在NAT10 OE hESCs中OCT4和SOX2的表达显著增加,而NANOG的表达没有明显改变。这些数据显示NAT10的过表达有助于促进hESCs的自我更新。
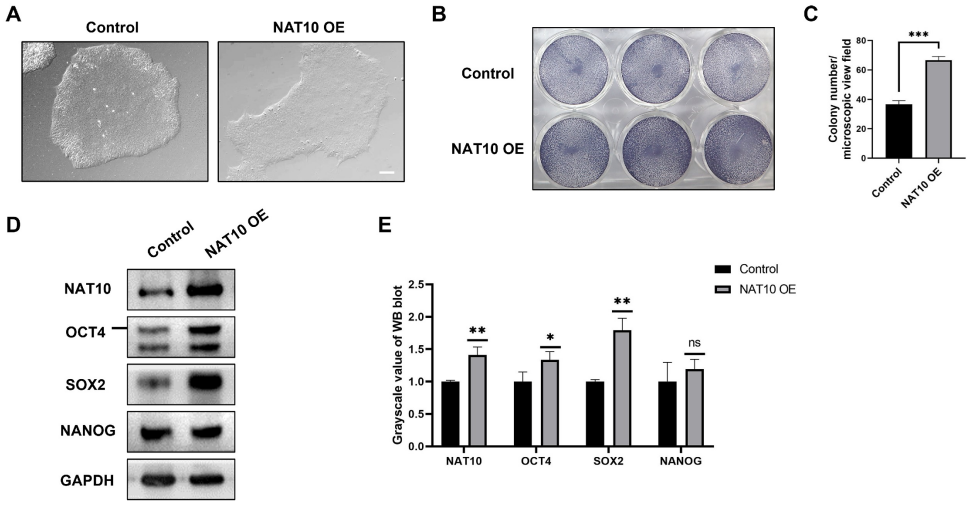
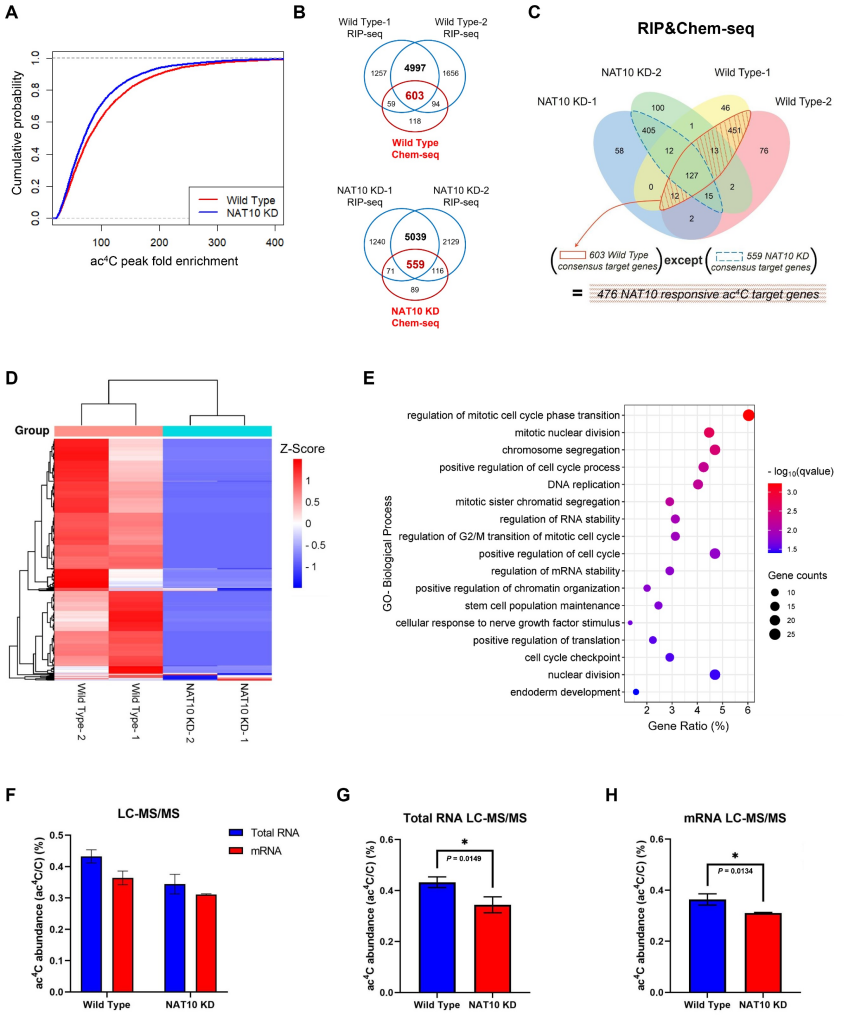
5、NAT10缺失导致靶基因上的ac4C缺失
研究在转录组规模上NAT10缺失对ac4C修饰的影响,作者对NAT10 KD hESCs进行了acRIP-seq实验,比较NAT10 KD hESCs与WT hESCs的ac4C峰和靶基因,在NAT10 KD hESCs中,ac4C乙酰化峰得分总体降低。作者又进行了基于NaCNBH3的化学ac4C测序,以消除假阳性,两种方法联合分析鉴定出476个NAT10应答的ac4C靶基因,热图还显示,在NAT10 KD的hESCs中,这些NAT10应答的ac4C靶基因的乙酰化水平显著降低。这些靶基因的GO-BP功能富集分析突出了几个功能组,如有丝分裂核分裂、DNA复制、细胞周期正调控、mRNA稳定性调控、干细胞群体维持和内胚层发育等,并且在这476个对NAT10应答的ac4C靶基因中,观察到了两个核心多能性调控因子,即OCT4和SOX2。
通过LC-MS/MS评估了WT和NAT10 KD hESCs的总RNA和mRNA中ac4C修饰水平。发现在hESCs中,mRNA上存在大量的ac4C修饰,与WT hESCs相比,NAT10 KD hESCs的ac4C丰度在总RNA和mRNA中都显著降低。这些结果表明,NAT10催化的ac4C乙酰化主要分布在mRNA上,这可能与hESC的细胞周期、mRNA稳定性和自我更新有关。
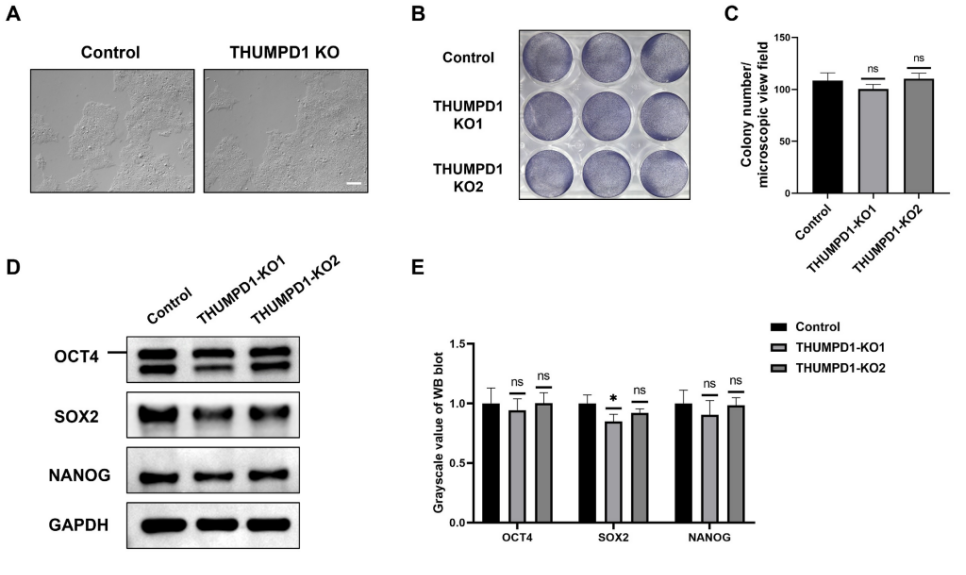
6、排除tRNA和rRNA的ac4C修饰对hESC自我更新的影响
tRNA ac4C乙酰化也依赖于关键蛋白辅助因子THUMPD1。为了进一步研究排除tRNA ac4C乙酰化对hESCs自我更新的重要贡献,作者通过CRISPR/Cas9靶向THUMPD1位点,构建了THUMPD1敲除(KO)的hESCs。形态学图像显示,THUMPD1 KO hESCs与对照组hESCs之间的细胞形态没有显著差异,并且两种hESCs形成的菌落数量也没有明显差异,说明THUMPD1敲除对hESCs的增殖率没有显著影响。通过Western blot检测THUMPD1 KO hESCs和对照组中OCT4、SOX2和NANOG的蛋白水平,与对照组hESCs相比,THUMPD1 KO hESCs中OCT4和NANOG的表达没有明显变化。同样,SNORD13是rRNA乙酰化所特别需要的,作者利用CRISPR/Cas9构建了SNORD13敲除(KO)的hESCs,与前面所述结果相似,与对照组相比,SNORD13 KO hESCs的细胞形态、菌落数量无显著差异,OCT4、SOX2和NANOG的表达也没有明显差异。上述结果说明rRNA和tRNA ac4C乙酰化对hESCs的自我更新没有产生显著影响。
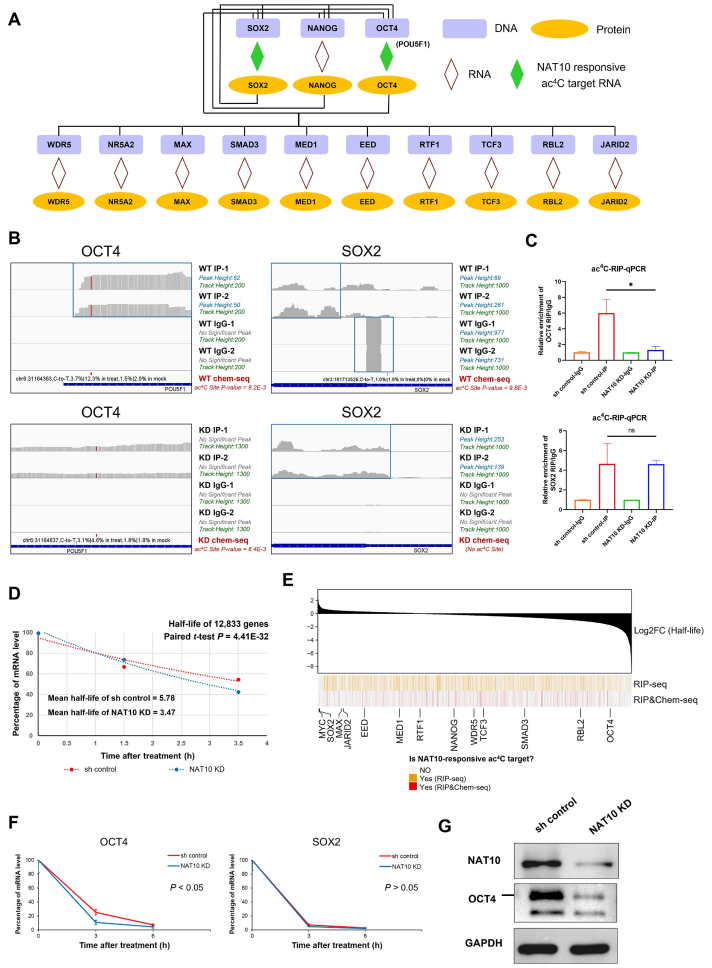
7、NAT10催化ac4C修饰靶向核心多能性因子OCT4,调控其mRNA的稳定性
为了进一步验证OCT4和SOX2的ac4C丰度,作者进行了acRIP-qPCR检测,并以IgG作为同型对照。结果显示,NAT10 KD细胞中OCT4的ac4C丰度明显降低,而与Sh对照组相比,SOX2没有明显变化,这表明SOX2对NAT10应答的ac4C靶基因的置信度较低。对NAT10 KD和sh对照的hESCs的mRNA半衰期分析得出,NAT10的扰动可能导致mRNA稳定性的整体显著下降,与sh对照相比,NAT10 KD中OCT4的半衰期下降最显著,SOX2没有显著下降。NAT10敲除后,OCT4 mRNA的稳定性显著降低,而SOX2 mRNA的稳定性没有显著变化。综上所述,这些结果表明核心多能性相关基因OCT4是NAT10介导的ac4C修饰的显著功能靶点,且NAT10维持了OCT4的mRNA稳定性和蛋白表达水平。
云序生物ac4C修饰研究六大模块
01 ac4C RNA修饰测序
对ac4C RNA乙酰化,目前最流行的检测手段为acRIP-seq技术,适用于ac4C RNA乙酰化谱研究,快速筛选ac4C RNA乙酰化靶基因。云序可提供mRNA和多种非编码RNA的ac4C测序:
ac4C 全转录组测序(涵盖mRNA,LncRNA,circRNA)
ac4C LncRNA测序(涵盖LncRNA和mRNA)
ac4C Pri-miRNA测序(涵盖Pri-miRNA和mRNA)
ac4C mRNA测序
ac4C rRNA测序
ac4C circRNA测序
ac4C tRNA测序
02 检测整体ac4C RNA修饰水平
精准高效,可以实现一次检测,9类修饰水平检测,一步到位。
03 ac4C RNA修饰上游酶的筛选
寻找上游直接调控ac4C RNA乙酰化的甲基转移酶。
04 ac4C RNA修饰靶基因验证
云序提供acRIP-qPCR服务,可针对mRNA,lncRNA,环状RNA等不同类型的RNA分子进行检测,低通量验证靶基因RNA修饰水平。
05 机制互作研究
筛选或验证RNA修饰直接靶点,研究RNA修饰靶基因的调控机制。
筛选或验证目标RNA互作基因或蛋白,研究相应的分子调控机制。
06 翻译组
检测转录本上核糖体的分布和翻译活性,了解蛋白质表达情况以及翻译过程。
云序生物服务优势
优势一:云序累计支持客户发表百余篇RNA修饰SCI论文,合计影响因子1000+, 其中RNA乙酰化测序有多达5篇影响因子接近20 分的高分论文。
优势二:累计完成数千例 RNA甲基化测序样本,全面覆盖医口、农口等各类样本。
优势三:全面检测mRNA和各类非编码RNA(circRNA,lncRNA,Pri-miRNA等)。
优势四:提供ac4C一站式服务:ac4C整体水平检测、acRIP-seq、acRIP-qPCR验证、RIP和RNA pull down等。
优势五:率先研发超微量acRIP测序技术,RNA量低至500ng起。
优势六:国内较全的RNA修饰测序平台,提供m6A、m5C、m1A、m7G、m3C、m6Am、O8G、ac4C乙酰化和2'-O-甲基化测序。
相关产品
往期回顾
云序用户农口IF20+论文:植物mRNA存在ac4C新型修饰,对RNA稳定性及翻译产生重要影响
云序用户1区,IF=23.168 | 多组学联合分析揭示胰腺癌中 RNA 乙酰化修饰的重要靶点
1区,IF=19.16| 云序RNA乙酰化测序acRIP-seq助力病毒复制机制研究!
1区,IF=16.8| 云序RNA乙酰化测序/acRIP-seq助力心梗机制研究!

上海云序生物科技有限公司
地址: 松江区莘砖公路518号18号2楼
网址: http://www.cloud-seq.com.cn
电话: 021-64878766
邮箱: market@cloud-seq.com.cn
